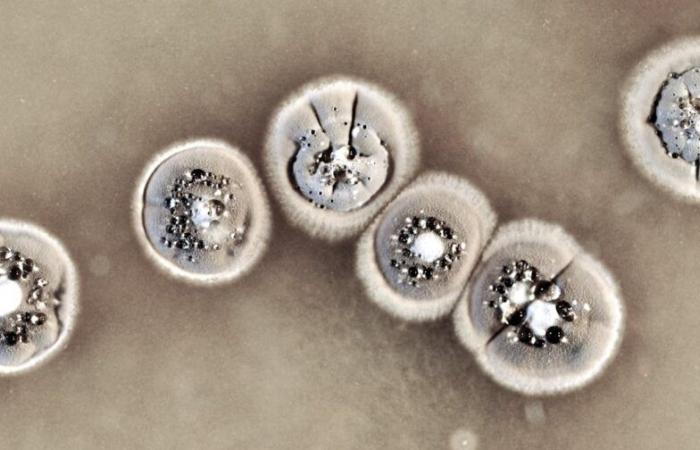
Certaines bactéries sont capables de produire des antibiotiques dans un contexte de compétition entre espèces, c’est le cas du genre Streptomyces. Une nouvelle découverte décortique la manière dont ils produisent ces molécules et ouvre la voie à la production de nouveaux antibiotiques et à la lutte contre la résistance aux antibiotiques.
La propagation de la résistance aux antibiotiques parmi les pathogènes humains et animaux est à l’origine d’une crise sanitaire contemporaine et d’une potentielle catastrophe future. L’Organisation mondiale de la santé (OMS) reconnaît la résistance aux antimicrobiens comme un problème socio-économique majeur et « l’une des dix plus grandes menaces pour la santé publique mondiale ». Il est désormais admis que l’usage massif et abusif des antibiotiques favorise l’émergence et la propagation de la résistance aux antimicrobiens chez les agents pathogènes humains. Une véritable course de vitesse est engagée : il est impératif d’innover pour identifier et produire de nouvelles molécules capables de contrer l’adaptation des bactéries à la pression des antibiotiques.
La principale Source de molécules naturelles pour l’utilisation des antibiotiques sont les bactéries du sol, notamment les Streptomyces. En effet, les bactéries établissent, grâce aux molécules qu’elles synthétisent, un équilibre avec les autres organismes colonisant le sol, dont la compétition qui consiste à éliminer la compétition.
Près d’un tiers de tous les antibiotiques connus proviennent de Streptomyces, ce qui place ce genre bactérien unique en deuxième position pour la découverte d’antibiotiques, juste derrière toutes les plantes supérieures.
Quand les bactéries jouent aux chimistes
Les streptomyces sont des bactéries non pathogènes qui comptent parmi les « chimistes » les plus compétents de la nature grâce à leur capacité à produire une grande diversité de métabolites spécialisés. Ces métabolites sont utilisés en médecine humaine et vétérinaire ainsi qu’en agronomie, notamment pour des molécules antibiotiques, antiprolifératives, antitumorales ou antioxydantes. Par exemple, la streptomycine produite par la bactérie Streptomyces griseusa été découvert comme antibiotique efficace contre la tuberculose par Selman Waksman, lauréat du prix Nobel de médecine en 1952.
Après l’âge d’or de la découverte des antibiotiques dans les années 1960 qui a anticipé la fin des maladies infectieuses, l’apparition de souches résistantes ne s’est pas accompagnée de la découverte et de la commercialisation de nouveaux agents thérapeutiques. La faute réside dans la forte probabilité de réisoler plusieurs fois une molécule déjà connue. Outre le fait que l’étendue de la diversité des molécules produites n’est pas connue, les processus (mécanismes et pressions) favorisant la diversité de ces molécules dans les organismes producteurs sont largement méconnus. Peut-on espérer bénéficier d’un renouvellement de notre arsenal antibiotique grâce à la capacité des producteurs à diversifier leurs gènes de biosynthèse ?
Au cours de la dernière décennie, les progrès des technologies de séquençage ont révélé que les génomes de Streptomyces regorgent d’un potentiel insoupçonné pour la synthèse de métabolites d’intérêt. En effet, alors que chaque souche était connue le plus souvent pour la synthèse d’une molécule révélée par son activité, leur génome comprend jusqu’à 40 groupes de gènes de biosynthèse (groupes de gènes biosynthétiquesBCG) représentant au total jusqu’à 10% de leur patrimoine génétique. Ce sont les organisations qui consacrent la majorité de leurs actifs à cette fonction. Chacun de ces BGC permet théoriquement la synthèse d’une nouvelle molécule. L’ère de la génomique venait de s’ouvrir avec ses espoirs et ses frustrations : espoirs d’une nouvelle diversité à explorer, frustrations de ne pas maîtriser les processus de création de diversité.
Le sol, véritable champ de bataille
La richesse et la diversité des antibiotiques produits par Streptomyces s’expliquent fondamentalement par leur rôle écologique pour la bactérie de son écosystème. Les Streptomyces vivent principalement dans les sols et particulièrement dans la rhizosphère, la zone du sol sous l’influence des racines de la plante, où se développe le microbiote végétal. Cet écosystème hétérogène aux conditions physico-chimiques changeantes est le siège d’une compétition féroce pour les nutriments, mais aussi de symbioses fertiles.
Les Streptomyces sont impliqués dans ces interactions biotiques multiples et complexes avec d’autres organismes vivant dans le sol (bactéries rhizosphériques, champignons, plantes et animaux) à travers les métabolites produits qu’ils synthétisent et excrètent (constituant l’interactome Streptomyces). Ces dialogues chimiques permettent l’équilibre des communautés vivant dans le sol.
Sous cette forte pression biologique, la course aux armements est permanente sous nos pieds pour assurer la production de nouvelles molécules actives garantissant la survie et la dissémination des bactéries. La résistance au médicament synthétisé par le producteur lui-même (afin d’éviter le suicide) doit évoluer comme un miroir de la capacité de biosynthèse. Nous avons montré qu’au sein d’une population de Streptomyces, alors qu’une seule souche produit l’antibiotique, les souches parentales non productrices sont résistantes, favorisant ainsi la dissémination du producteur et de ses apparentés. Ce faisant, les producteurs sont aussi un réservoir de résistances en devenir (on parle de « proto-résistance », ancêtre de la résistance identifiée chez nos pathogènes).
Des échanges génétiques colossaux
Au laboratoire DynAMic, nous avons récemment montré que les Streptomyces sont capables d’échanger de grandes quantités d’ADN au contact les uns des autres. En effet, ces transferts de gènes ou transfert horizontal se produisent lors de la conjugaison entre souches originaires de la rhizosphère. Le génome de Streptomyces est riche en éléments génétiques mobiles appelés AICE, pour Actinomycètes éléments intégratifs et conjugatifs (élément conjugatif et intégrateur des actinomycètes), qui favorisent, outre la mobilité de leur propre ADN génomique (entre 20 et 40 kilobases), celle de l’ADN chromosomique entre les partenaires impliqués dans le croisement.
Lors d’un seul croisement, le transfert d’un ou plusieurs de ces AICE s’accompagne de celui de plusieurs fragments d’ADN représentant au total jusqu’à 30 % du patrimoine génétique du parent donneur.
Fourni par l’auteur
Le génome des Streptomyces utilisés dans cette expérience comprenant environ 12 millions de paires de bases, cela représente plusieurs centaines, voire milliers de kilobases, et donc autant de gènes (1 gène ≃ 1 000 bases chez les bactéries) ! Assez pour que des voies biosynthétiques entières soient transférées ; les gènes nécessaires à la synthèse et à la résistance au médicament produit étant regroupés en régions d’une centaine de kilobases environ.
Une fois chez le parent receveur, l’ADN du donneur insère et remplace les informations présentes sur le chromosome et entraîne des innovations génétiques. Ainsi, nous avons démontré que plus de 90 % des descendants obtenus entre une bactérie donneuse AICE et une bactérie receveuse présentent un arsenal de biosynthèse de métabolites distinct des deux parents ; les descendants ont acquis ou perdu des voies de biosynthèse entières, ou ont combiné deux voies pour en générer une nouvelle.
Ce nouveau phénomène assure un brassage génétique, comparable à celui se produisant lors de la méiose (division cellulaire permettant la formation de gamètes) des organismes à reproduction sexuée. Ce mélange induira des altérations drastiques du métabolisme spécialisé conduisant à de nouvelles variantes d’antibiotiques qui faciliteraient l’adaptation des populations de Streptomyces à leur écosystème. Le réarrangement des BGC pourrait jouer un rôle dans la « guerre bactérienne » intra- et interspécifique. À des échelles de temps plus longues, c’est cette pression qui pourrait favoriser la divergence des espèces bactériennes en les spécialisant vers un écosystème spécifique. On sait que la recombinaison génétique influence considérablement le processus de divergence ou de spéciation.
Cette découverte ouvre de nouvelles perspectives pour la biotechnologie et la médecine. En plus de permettre de mieux comprendre les mécanismes de transfert et de recombinaison de l’ADN afin de mieux comprendre l’évolution et l’adaptation des bactéries du sol, elle permet d’exploiter ce phénomène pour diversifier les gènes et groupes de gènes de biosynthèse de métabolites spécialisés. Cela facilite la découverte de nouvelles biomolécules et pourrait accélérer la lutte contre la résistance bactérienne en fournissant une Source constante de nouveaux antibiotiques. Le bricolage évolutionniste, cher à François Jacob, nous surprend en fabriquant sans cesse du nouveau à partir de ce qui existe déjà, offrant ainsi une opportunité sans cesse renouvelée de découvrir de nouveaux composés bioactifs, dont des antibiotiques, essentiels dans la guerre contre les pathogènes.